CrySPYの使い方
CrySPYは、結晶構造の探索ソフトです。 第41回CMDワークショップのマテリアルズ・インフォマティクスコースでCrySPY講義・演習が行われます。 また共立出版からマテリアルズインフォマティクスという教科書が発売され、その第3章が「結晶構造探索ツールCrySPY」との事なので、自習も可能なようです。
この文章では CrySPY 0.10.4 を VirtualBox の上で動作する Ubuntu 20.04 にインストールする方法を書きます。 Anaconda をインストールするとディスク容量をかなり消費するようです。 Virtual Box のデフォルトのディスク容量は 10 GB となっていますが、もっと大きくする必要がありました。 とりあえず 40 GB で動いています。 メモリは(いくつ必要なのかは分かりませんが) 8192 MB (8 GB) としました。
CrySPYのインストール
インストール:: CrySPYの手順に従って、以下のインストールを行います。
- Python環境
- CrySPY本体
- soiap(構造最適化ソフト)
0. 前準備
まず、なにはともあれ諸々のアップデートをしておきます。ターミナルに以下を打ち込んで、アップデートと再起動をしておきます。
sudo apt -y update && sudo apt -y dist-upgrade && sudo apt -y autoremove && sudo reboot
そして apt で必要そうなものをインストールしておきます。
既にインストールしてあるものがあっても気にせずそのままコピペします。
sudo apt -y install git gfortran build-essential emacs libatlas3-base libatlas-base-dev liblapack3 tree1. Python環境構築
CrySPY 0.10.4 のシステム要件としてPython本体と3つのライブラリをインストールする必要があるようです。
1.1 Anaconda (Python) のインストール
Python の実行環境として Anaconda をインストールします。 詳しいインストールの仕方はLinux版Anacondaのインストールを参考にしてください。
cd ~ wget https://repo.anaconda.com/archive/Anaconda3-2022.05-Linux-x86_64.sh bash ./Anaconda3-2022.05-Linux-x86_64.sh
インストールが完了したら一旦ターミナルを閉じ(sshならログアウトし)、ターミナルをもう一度起動します。
インストールに成功していれば、ターミナルの左上に (base) という文字が追加されているはずです。
1.2 pymatgenのインストール
pymatgenをInstallationにしたがってインストールします。
Conda と PipによるとPythonのパッケージインストールは pip というコマンドを使う方法と conda というコマンドを使う方法があるようです。
なんだかよく分からないですが、インストール方法のページでは conda を試した後で pip を試すように書いている???
conda の方はぐるぐる回るだけでいつまでたってもインストールできなかったのでpipの方でインストールします。
conda install --yes numpy scipy matplotlib
pip install pymatgen
なんか、普段使いのPCクラスタにインストールしようとしたらERROR: Cannot uninstall 'ruamel-yaml'. It is a distutils installed project and thus we cannot accurately determine which files belong to it which would lead to only a partial uninstall.って怒られた。
ググったら無視する方法が載っていたので、真似して pip install pymatgen --ignore-installed ruamel.yaml としたが、動いて無さそう…。
良く分からないですが Anaconda パッケージ アップデート方法 を見ながら Anaconda 本体のアップデート conda update -n base conda とか色々やったら動くようになりました。
なぜ動くようになったのかは分かりません…Pythonなんにも分かりません…。
1.3 PyXtalのインストール
PyXtal Installationの通りにインストールします。
pip install pyxtalとすると、以下のエラーが出ましたが
ERROR: pip's dependency resolver does not currently take into account all the packages that are installed. This behaviour is the source of the following dependency conflicts.
daal4py 2021.5.0 requires daal==2021.4.0, which is not installed.その後で
Successfully installed ase-3.22.1 llvmlite-0.39.0 numba-0.56.0 numpy-1.22.4 pooch-1.6.0 py3Dmol-1.8.0 pyshtools-4.10 pyxtal-0.5.0とも言われていて、なんとなく動いている様です…。
1.4 comboのインストール
combo3 Installのとおり git を利用してパッケージをダウンロードしたのちにインストールします。
cd ~/
git clone https://github.com/tsudalab/combo3.git
cd combo3
python setup.py install2. CrySPYのインストール
2.1 CrySPY本体のインストール
mkdir ~/CrySPY_root
cd ~/CrySPY_root
git clone https://github.com/Tomoki-YAMASHITA/CrySPY.git CrySPY-0.10.42.2 CrySPYの実行スクリプトの作成
この手順は、チュートリアルではテスト計算の部分で出てくるものですが、ここでやっておきます。
まずは PATH が通っているディレクトリを準備しておきます。
ここでは ~/bin/ を作って PATH を通す例を書きます。
ホームディレクトリに bin/ ディレクトリを作り、その中に cryspy という名前のシェルスクリプトを作成します。
この cryspy というシェルスクリプトが、CrySPYの実行スクリプトになります。
cd ~/
mkdir bin
cd bin/
emacs -nw cryspy
シェルスクリプト cryspy の中身は以下の通りです。
#!/bin/sh
python3 -u ~/CrySPY_root/CrySPY-0.10.4/cryspy.py 1>> log 2>> err
CrySPYをインストールしたディレクトリ名のバージョンを間違わないようにしてください。
cryspy スクリプトを作成したら、実行権限を付けておきます。
なお CrySPY の新しいバージョンが公開されたときには CrySPY_root/ の下に別バージョンのディレクトリを新しく作り、この実行スクリプトのパスを編集することによって、使いたい方のバージョンを選択します。
chmod +x cryspy
次に ~/bin/ に PATH を通します。
具体的には ~/.bashrc に export PATH=$PATH:$HOME/bin を追記したのち source コマンドで有効化します。
cd ~/
emacs -nw ~/.bashrcexport PATH=$PATH:$HOME/binsource ~/.bashrc
~/bin/ は ecalj をインストールすると作成されるディレクトリなので、私はそれを再利用しています。
2.3 cal_fingerprintのコンパイル
ベイズ最適化を行う場合はインストールを行う必要がある。
チュートリアルには Makefile の編集が手順に入っていますが gfortran でコンパイルする場合は、編集の必要は無さそうです。
cd ~/CrySPY_root/CrySPY-0.10.4/CrySPY/f-fingerprint/ make ls cal_fingerprint
Intel Fortran Compilerなど別のコンパイラを持っていて使いたい場合は make を実行する前に Makefile を編集して使用するコンパイラを指定する模様。
コンパイルに成功していれば cal_fingerprint という名前の実行ファイルが出来ているはずです。
2.4 CrySPYのユーティリティのインストール
CRYSPYユーティリティ (オプション)にしたがって、ユーティリティをインストールします。
ローカルPCにインストールする事になっていますが、サーバーの方に入れておいてもいいでしょう。
git で導入して、ユーティリティディレクトリを PATH に追加しておきます。
cd ~/CrySPY_root/
git clone https://github.com/Tomoki-YAMASHITA/CrySPY_utility.git
cd ~/
emacs -nw ~/.bashrcexport PATH=$PATH:$HOME/CrySPY_root/CrySPY_utilitysource ~/.bashrc3. soiap(構造最適化ソフト)のインストール
次にsoiapのインストールを行います。
これは、構造最適化ソフトの1種で、どうやらCrySPYでは、最も簡単な構造最適化ソフトという位置づけの様です。
構造最適化ソフトとして、最終的にQuantum ESPRESSOやLAMMPSを使う予定だとしても、テスト計算はsoiapでやるのが良いみたいなので、インストールを行います。
cd ~/
git clone https://github.com/nbsato/soiap.git
cd ~/soiap/src/
make4. テスト計算: soiapを用いたSiの構造予測
soiapを用いたテスト計算を行います。
具体的には、チュートリアルのsoiap in your local PCの手順をそのまま写しただけです。
4.1 入力ファイルの準備
CrySPY + soiap で構造最適化を行う際に必要な入力ファイルは3種類あります。
- CrySPYの入力ファイル: cryspy.in
- ジョブスクリプト: calc_in/job_cryspy
- soiapの入力ファイル: calc_in/soiap.in_1
まず、適当に練習用のディレクトリを作り移動します。
cd ~
mkdir project
cd project
mkdir practice
cd praciticeサンプルプロジェクトをコピーしてきます。
cp -r ~/CrySPY_root/CrySPY-0.10.4/example/soiap_Si8_RS/ .
cd soiap_Si8_RS/
中身を確認すると CrySPY そのものへの入力ファイルである cryspy.in と soiap 関連のファイルが入っている calc_in/ ディレクトリが見つかります。
calc_in/ ディレクトリの中には CrySPY から soiap を呼び出すためのシェルスクリプト(ジョブスクリプト)ファイルである job_cryspy と soiap の入力ファイルである soiap.in_1 が存在しています。
4.1.1 CrySPY本体の入力ファイル: cryspy.in
まずは cryspy.in の編集を行います。
emacs -nw cryspy.in[basic]
algo = RS
calc_code = soiap
tot_struc = 5
nstage = 1
njob = 2
jobcmd = bash
jobfile = job_cryspy
[structure]
natot = 8
atype = Si
nat = 8
[soiap]
soiap_infile = soiap.in
soiap_outfile = soiap.out
soiap_cif = initial.cif
[option]
カギ括弧 [] で囲まれたものがセクションの名前です。
上記のcryspy入力ファイルには basic, structure, soiap, option という4つのセクションが存在していることが分かります。
[basic]セクションの中の jobcmd が、デフォルトでは zsh になっているので bash を指定するように編集します。
4.1.2 ジョブスクリプト: calc_in/job_cryspy
次に calc_in/job_cryspy の編集を行います。
emacs -nw calc_in/job_cryspy#!/bin/sh
# ---------- soiap
EXEPATH=~/soiap/src/
$EXEPATH/soiap soiap.in 2>&1 > soiap.out
# ---------- CrySPY
sed -i -e '3 s/^.*$/done/' stat_job
EXEPATH の部分に soiap のインストールディレクトリのパスを書きます。
このページの通りにインストールしていれば EXEPATH=~/soiap/src/ となるはずです。
4.1.3 soiapの入力ファイル: calc_in/soiap.in_1
最後の入力ファイルは soiap への入力ファイルである calc_in/soiap.in_1 です。
こちらは編集する必要がなく、中身を眺めるだけです。
crystal initial.cif ! CIF file for the initial structure
symmetry 1 ! 0: not symmetrize displacements of the atoms or 1: symmetrize
md_mode_cell 3 ! cell-relaxation method
! 0: FIRE, 2: quenched MD, or 3: RFC5
number_max_relax_cell 100 ! max. number of the cell relaxation
number_max_relax 1 ! max. number of the atom relaxation
max_displacement 0.1 ! max. displacement of atoms in Bohr
external_stress_v 0.0 0.0 0.0 ! external pressure in GPa
th_force 5d-5 ! convergence threshold for the force in Hartree a.u.
th_stress 5d-7 ! convergence threshold for the stress in Hartree a.u.
force_field 1 ! force field
! 1: Stillinger-Weber for Si, 2: Tsuneyuki potential for SiO2,
! 3: ZRL for Si-O-N-H, 4: ADP for Nd-Fe-B, 5: Jmatgen, or
! 6: Lennard-Jones
今回は触りませんが、例えば external_stress_v に同じ値を入れれば、静水圧をかけられそうな感じですね。
4.2 CrySPYの実行
入力ファイルの準備が整ったので、いよいよ CrySPY を実行します。 すでに計算ディレクトリにいるとおもいますが、一応確認しておきます。
cd ~/project/practice/soiap_Si8_RS実行は「2.2 CrySPYの実行スクリプトの作成」で作ったシェルスクリプトを計算ディレクトリから呼ぶだけです。
cryspy公式のチュートリアルのFirst runでは & を付けてバックグラウンド実行をしていますが、別にフォアグラウンドで実行してもよいでしょう。
終了後に ls でファイルを確認すると、以下のような感じになっているはずです。
calc_in cryspy.in cryspy.out cryspy.stat data err log
失敗していると cryspy.out や cryspy.stat は作成されず err や log が出来ているかもしれません。
その場合は cat err としてエラーメッセージを確認すると何か分かるかも…
まず log の確認をします。
cat logすると、以下のようになっていました。
2022/08/07 14:08:26
CrySPY 0.10.3
Start cryspy.py
Read input file, cryspy.in
Write input data in cryspy.out
Save input data in cryspy.stat
# --------- Generate initial structures
Structure ID 0 was generated. Space group: 3 --> 3 P2
Structure ID 1 was generated. Space group: 159 --> 159 P31c
Structure ID 2 was generated. Space group: 112 --> 112 P-42c
Structure ID 3 was generated. Space group: 168 --> 168 P6
Structure ID 4 was generated. Space group: 98 --> 98 I4_1220番から4番まで、5種類の結晶構造がランダムに生成されていることが分かります。 なお、公式チュートリアルでは、以下のようになっており、実行するたびに生成される結晶構造が異なることが分かります。
2020/11/11 19:59:18 CrySPY 0.9.0 Start cryspy.py Read input file, cryspy.in Write input data in cryspy.out Save input data in cryspy.stat # --------- Generate initial structures Structure ID 0 was generated. Space group: 136 --> 139 I4/mmm Structure ID 1 was generated. Space group: 98 --> 98 I4_122 Structure ID 2 was generated. Space group: 16 --> 16 P222 Structure ID 3 was generated. Space group: 36 --> 36 Cmc2_1 Structure ID 4 was generated. Space group: 36 --> 36 Cmc2_1
次に cryspy.stat の中身を確認します。
[basic]
algo = RS
calc_code = soiap
tot_struc = 5
nstage = 1
njob = 2
jobcmd = bash
jobfile = job_cryspy
~(中略)~
[status]
id_queueing = 0 1 2 3 4
色々な情報が書かれていますが、重要なのは最後の方に書かれている id_queueing = 0 1 2 3 4 です。
番号は、それぞれ作成された結晶構造のIDに対応しています。
そして、このキューに残っている結晶構造は、まだ実際には計算されていません。
つまり cryspy の最初の実行では、初期構造を作成しているだけです。
そこで、もう一度 cryspy を実行してみます。
cryspy
二度目の実行が終わったら、もう一度 log や cryspy.stat を確認します。
以下は cryspy.stat の最後の方の部分です。
[status]
id_queueing = 2 3 4
id 0 = Stage 1
id 1 = Stage 1
0番と1番の結晶構造がStage 1になり、残りの2-4番がキューの中に残っていることが分かります。
もう一回 cryspy を実行し cryspy.stat の最後を確認します。
cryspy
tail -n 5 cryspy.stat[status]
id_queueing = 4
id 2 = Stage 1
id 3 = Stage 14番だけがキューに残っています。 もう一回。
cryspy
tail -n 5 cryspy.stat[status]
id_queueing =
id 4 = Stage 1
キューに溜まっていた結晶構造が全て無くなり、4番の構造がStage 1です。
もう一回 cryspy を実行します。
[status]
id_queueing =
キューにもStage 1にも、何もなくなりました。
この時点でのそれぞれの構造のエネルギーを比較します。
この情報は data/cryspy_rslt_energy_asc に書き込まれています。
cat data/cryspy_rslt_energy_asc Spg_num Spg_sym Spg_num_opt Spg_sym_opt E_eV_atom Magmom Opt
1 159 P31c 9 Cc -3.826929 NaN not_yet
0 3 P2 3 P2 -3.746252 NaN not_yet
2 112 P-42c 111 P-42m -2.849364 NaN not_yet
4 98 I4_122 5 C2 -2.518968 NaN not_yet
3 168 P6 3 P2 10.084604 NaN not_yetこの中では1番の結晶構造が最もエネルギーが低く安定です。 これらの構造をCIFに書き出してVESTAで描画してみます。 CrySPY Utility の extract_struc.pyのページにCIFの出力方法が書いてあるのでそれに従います。 下記のコマンドで、構造最適化後の全ての結晶構造をCIFに書き出します。
extract_struc.py data/pkl_data/opt_struc_data.pkl -as下記の図は、今回計算した5種類の構造の中で最も安定な構造であるID番号1番の物です。
なのですが、5種類の構造の中でこの構造だけVESTAで開こうとするとVESTAが落ちました…。 仕方ないので、出力されたCIFファイルの中の数値を読みながら自分でVESTAに打ち込んだやつです。
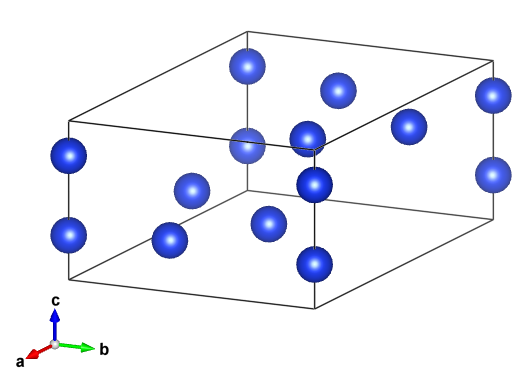
シリコンの安定な結晶構造はダイヤモンド構造であるべきです。 しかしながら、見ての通り上記の結晶構造はダイヤモンド構造ではありません。 たった5種類しか調べていないので、これは当然のことです。
そこで cryspy.in のなかの tot_struc の数字を大きくして、たくさんの構造を調べます。
例えば tot_struc = 10 などとして cryspy を繰り返せば続きから計算できます。
別の計算機で20種類の構造を計算しました。 偶然ですが、IDが19番の構造が我々の良く知るいわゆる普通のダイヤモンド構造になりました。
Spg_num Spg_sym Spg_num_opt Spg_sym_opt E_eV_atom Magmom Opt
19 215 P-43m 227 Fd-3m -4.336409 NaN done
1 87 I4/m 2 P-1 -4.060244 NaN not_yet
9 100 P4bm 99 P4mm -4.052860 NaN done
3 148 R-3 148 R-3 -4.014984 NaN not_yet
0 230 Ia-3d 230 Ia-3d -3.996916 NaN done
2 142 I4_1/acd 230 Ia-3d -3.996730 NaN not_yet
5 225 Fm-3m 225 Fm-3m -3.880172 NaN done
6 123 P4/mmm 123 P4/mmm -3.801159 NaN not_yet
15 52 Pnna 52 Pnna -3.732424 NaN not_yet
4 187 P-6m2 187 P-6m2 -3.544994 NaN done
7 194 P6_3/mmc 63 Cmcm -3.535674 NaN not_yet
11 127 P4/mbm 127 P4/mbm -3.309458 NaN not_yet
14 22 F222 5 C2 -3.302189 NaN not_yet
12 123 P4/mmm 123 P4/mmm -3.000849 NaN done
16 155 R32 5 C2 -2.225820 NaN not_yet
18 139 I4/mmm 139 I4/mmm -2.114425 NaN done
10 166 R-3m 166 R-3m -1.870017 NaN not_yet
17 221 Pm-3m 221 Pm-3m -1.757427 NaN done
13 100 P4bm 100 P4bm -1.604391 NaN not_yet
8 227 Fd-3m 166 R-3m -0.564718 NaN not_yet